来自Bruker的TopSpin™软件包是为NMR数据分析和NMR光谱的采集和处理而设计的。这个软件包,TS3.5pl6是Bruker的最新版本,具有新的,易于使用的GUI,它提供了方便访问大量的实验库,包括典型的Bruker脉冲序列,以及为学术和工业用户生成的实验库。
通过简单的拖放,用户可以构建和组织自己定制的实验库,支持基于脚本的参数修改,为实验设置和优化提供了多种选项,使高级实验的设置简单高效。
TopSpin提供了一个完全面向工作流的用户界面,并利用现代Windows / MacOS/ CentOS操作系统的现代64位特性来优化内存使用。其新的面向工作流和直观的用户界面和优化的样本数据管理都提供了一长串额外的创新功能,意味着专门加快操作和样本分析吞吐量,以获得更好的成本效率。如果需要帮助教育和学术界机构改善他们的学生课程,TopSpin也提供学生版本。
TopSpin是用一个非常直观的界面构建的,它应用了字处理、表示程序或图形所熟悉的最广泛的标准,为Windows上的NMR应用程序提供了相同的外观和感觉®, Mac和Linux®.
特性
TopSpin™软件包的主要特点如下:
- 完全控制Avance和傅里叶光谱仪
- 综合功能的处理,显示和分析多维和单光谱
- 直观的行业标准用户界面,是普通用户和专家的理想解决方案
- 无限离线处理能力
- 协助收购
- 先进的非均匀采样技术
- 小分子特征
- BioToolsTM -简单的生物分子核磁共振
- 方法的开发环境
- 用CMC-seTM简单有效地阐明了微小分子的结构
- 固态核磁共振线形分析,包括动态核磁共振
- 结果发布,预定义和用户定义的布局
- 法规遵从性支持工具(审计跟踪、自动归档、电子签名)
- 大学和学生专用许可证
技术细节
用户界面
- 完整的处理、显示和分析多维和单光谱功能
- 直观的行业标准用户界面,标准用户和专家的理想解决方案
- 协助收购
- 反褶积/光谱模拟迭代
- 结果发布,预定义和用户定义的布局
- 个人用户定制(实验、工具栏、命令、工作流)
- 完整的文档
- 法规遵从性(GMP, GLP)支持工具(电子签名,审计跟踪,自动归档)
- PC (Linux / Windows)或Mac
- 为各种应用程序提供灵活的许可
- 专为大学生和
支持操作系统
支持光谱仪计算机上的TopSpin 3.5:
- Windows 7(64位)
- CentOS 7.1(64位)
- CentOS 5(64位)
支持数据站的TopSpin 3.5:
- Windows Vista(32位)
- Windows 8.1(32位和64位)
- Windows 7(32位和64位)
- CentOS 7.1(64位)
- CentOS 5(32位和64位)
- Mac-OS X 10.8或更新版本
为了在CentOS下有效安装和操作TopSpin,需要额外的软件包:
- CentOS 7安装要求
- CentOS 5的TopSpin安装要求
采购和处理包
- BioTools™-蛋白核磁共振简化
- 固体lineshape分析
- 非均匀采样(选项)
- 小分子结构说明(选项)
- 结构化学位移预测(选项)
- 结构置信度分析(选项)
应用程序
绝对量化- CMC-q
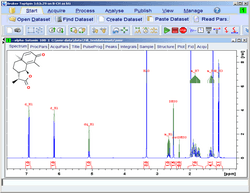
它可以对一系列样品进行自动绝对定量,从而使用户能够定义物质的质量含量、纯度和相对数量。这在药物发现筛选应用中提供了完全自动化的浓度测定。
结构说明CMC-se
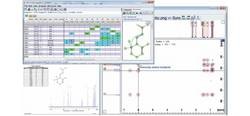
从一个化学公式和标准的核磁共振实验开始,峰被自动选择并进入一个相关表。然后使用结构确定算法解释这些数据,并生成与数据一致的结构。然后,这些结构可以与化学位移预测的比较进行排名,从而缩小可能性,以帮助研究人员建立它们的结构。
非均匀采样
TopSpin 3.0目前具有非均匀采样(NUS),这是一个完全集成的采集和处理功能。它允许重复使用,可用于各种核磁共振实验。一个自动生成和优化的NUS稀疏列表建立抽样模式,导致分数数据采集。然后,多维分解允许计算缺失的数据点,使完整数据集的常规傅里叶变换处理,所有这些都以完全自动的方式执行。
新加坡国立大学的障碍消除了时间分辨率多维核磁共振光谱,和特别有利于多维生物分子核磁共振实验,节省了时间的因子4在3 d实验或10甚至更多的4 d和5 d实验可以实现。新的蛋白质结构测定实验在NUS中是可行的。
基于二维光谱的小分子应用也可以获得更快的捕获因子2。这里的一个关键亮点是,NUS在不增加总体采集时间的情况下实现了光谱分辨率和质量的改善。
新加坡国立大学在TopSpin的实施是与Orekhov教授和同事的研究伙伴关系的结果。NUS在TopSpin 3.0中受益于完善的算法和程序,如NUS- sampler和MDDNMR (Orekhov, V.Y., I. Ibraghimov, and M. billette, J. Biomol)。核磁共振,2003年。27 (2): p。165 - 173年)
动力中心
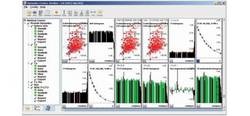
新的动态中心是一个面向技术的工作流,利用高效的蛋白质数据评估,提供所有适用的动态参数,如Rex, T1, T2,用户交互最少。这种新能力主要是为了探索大型生物分子的动力学。